Forschung
Max-Planck-Forschungsgruppe Tessarz
Chromatin besteht aus DNA und Proteinen und ist im Zellkern zu finden. Wir wollen verstehen, wie die Architektur des Chromatins die Genexpression, also die Aktivität von Genen, regulieren kann. Insbesondere wollen wir aufklären, wie altersbedingte Veränderungen im Epigenom die Transkription und letztlich die Entscheidungen über das Zellschicksal beeinflussen. In den letzten Jahren hat uns die Verbindung anderer zellulärer Wege mit epigenetischen Mechanismen fasziniert und wir haben unsere Forschung erweitert, um den Zusammenhang zwischen Stoffwechsel und Kommunikation zwischen Organellen auf die Chromatinstruktur zu untersuchen. Zu diesem Zweck kombinieren wir mechanistische Ansätze in der Bäckerhefe S. cerevisiae und etablierten Zellkultursystemen mit der Analyse von Primärzellen und Geweben unter Verwendung von Biochemie, Zellbiologie und einer Vielzahl modernster Deep Sequencing-Technologien.
Ausgewählte Projekte
Chromatin-vermittelte Regulierung der Genexpression
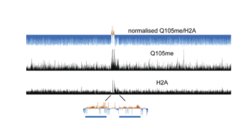
Wir haben kürzlich eine Histonmodifikation (Glutamin-Methylierung von Histon H2A an Glutamin 105) identifiziert, die ausschließlich mit der Funktion der RNA-Polymerase I verbunden ist. Diese Modifikation reguliert die Zugänglichkeit der rDNA, indem sie die Bindung des Histon-Chaperons FACT hemmt, was zu einem verminderten Einbau von H2A in den rDNA-Locus führt (Tessarz et al., 2014). Anknüpfend an diese Arbeit identifizierten wir spezifische Proteine, welche diese Modifikation auslesen und mit der Ribosomen-Assemblierung, der rRNA-Prozessierung und der Erkennung von Stoffwechselzuständen verbunden sind. Interessanterweise hängt die Methylierung von H2AQ105me von einer anderen Histonmodifikation ab, der Acetylierung von Lysin 56 in Histon H3, einer Modifikation, die - neben anderen - mit der rRNA-Transkription in Verbindung gebracht wurde. Dieser Forschungsstrang führte auch zur Identifizierung eines sehr interessanten zellulären Crosstalks. Mutationen in H3K56 führen zu Veränderungen in der naszierenden RNA-Transkription, während die RNA-Spiegel im Steady-State nahezu unbeeinflusst bleiben. Durch einen genomweiten Screen nach genetischen Interaktoren von H3K56A identifizierten wir das RNA-bindende Protein Puf5. Wir konnten zeigen, dass Puf5 Transkripte kontextabhängig entweder stabilisieren oder abbauen kann und die regulären mRNA-Spiegel aufrechterhält, indem es die Chromatin-vermittelte Regulation der naszierenden Transkription mit mRNA-Abbauwegen verbindet (Kochan et al., 2021). Neben unserer Arbeit zur Histonmodifikation und ihrer Rolle bei der Regulation der Genexpression sind wir auch sehr daran interessiert zu verstehen, wie die Chromatinarchitektur die Transkription direkt regulieren kann. Ein Beispiel ist unsere Arbeit über das Histon-Chaperon FACT und wie es den Zugang zu Promotoren reguliert. In embryonalen ES-Zellen hemmt FACT die Antisense-Transkription, indem es das Nukleosom -1 aufrechterhält und damit den Zugang zum Promotor einschränkt (Mylonas et al., 2018). Derzeit erweitern wir diese Arbeit, um zu verstehen, wie Chromatinarchitektur und Transkriptionsregulation im Kontext der Alterung zusammenhängen.
Verbindung von Stoffwechsel und Epigenom
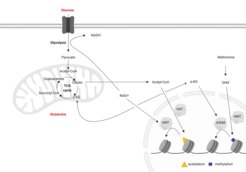
Zentrale Metabolite wie Acetyl-CoA, S-Adenosylmethionin oder das NAD/NADH-Verhältnis sind eng mit epigenetischen Reaktionen verknüpft, da sie als Substrate oder Cofaktoren für Enzyme dienen, die Histone und DNA modifizieren. Umgekehrt steht auch die Induktion oder Repression von Stoffwechselenzymen unter epigenetischer Kontrolle. Da das Altern mit Veränderungen sowohl im Stoffwechsel als auch im Epigenom einhergeht, möchten wir verstehen, wie die Verbindung zwischen diesen beiden essentiellen zellulären Wegen beeinflusst wird. Wir verwenden mesenchymale Stammzellen (MSCs), die aus dem Endosteum von Mäusen stammen, um dieses Zusammenspiel zu untersuchen. MSCs sind wichtig für die Aufrechterhaltung der Integrität des Knochens, sind aber auch essentiell für die Hämatopoese und spielen somit eine kritische Rolle während des Alterungsprozesses. Aus früheren Arbeiten wissen wir, dass sich MSCs in Chondrozyten, Osteoblasten und Adipozyten differenzieren können, um Knochen, Knorpel und Fett im Knochen zu regenerieren. Mit zunehmendem Alter wird diese Differenzierung jedoch in Richtung der adipogenen Linie verzerrt. Wir konnten zeigen, dass diese Defekte in der Osteogenese von altersassoziierten Veränderungen im Chromatin ausgehen, die auf Veränderungen der Histon-Acetylierungszustände in gealterten MSCs zurückzuführen sind (Pouikli et al., 2020). Diese Veränderung der Histon-Acetylierung ist eine Folge des beeinträchtigten Acetyl-CoA-Exports aus den Mitochondrien ins Zytoplasma aufgrund geringerer Spiegel des für diese Reaktion verantwortlichen Transporters (CiC). Wir konnten zeigen, dass die Abnahme von CiC von einer Zunahme der mitochondrialen Qualitätskontrollmechanismen abhängt, die zu einer höheren Umsatzrate des Transporters in gealterten Zellen führen. Auffallend ist, dass die Umgehung des Acetyl-CoA-Exports durch die Supplementierung des Mediums mit Acetat die Histon-Acetylierung und die osteogene Differenzierungsfähigkeit gealterter MSCs wiederherstellt, was das metabolisch-epigenetische Zusammenspiel in den Mittelpunkt der Differenzierungsveränderungen stellt. Zusammen mit Arbeiten aus anderen Laboren zeigt dies, dass Eingriffe auf epigenetischer Ebene ein Weg zur zellulären Verjüngung sein könnten.
Epigenetische Heterogenität

Ein Merkmal der Alterung ist eine Zunahme des biologischen Rauschens in vielen Geweben. Um dies auf der Ebene des Chromatins zu untersuchen und um zu verstehen, ob alle Zellen in unseren Geweben in gleicher Weise betroffen sind, setzen wir epigenomische Einzelzellkartierungen ein.
Darüber hinaus wollen wir verstehen, ob das potentielle Rauschen in in vitro Systemen erhalten werden kann. Um dieses potenzielle Phänomen des epigenetischen Gedächtnisses zu untersuchen, verwenden wir organoide Systeme, die wir direkt aus gealtertem Gewebe aufbauen.


