Forschung
Assoziierte Forschungsgruppe Rorbach
Ausgewählte Projekte
1. Mitochondriale Proteinsynthese
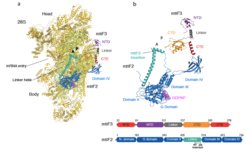
Die Translationsinitiation in menschlichen Mitochondrien beruht auf spezialisierten Mitoribosomen und Initiationsfaktoren, mtIF2 und mtIF3, die sich von ihren bakteriellen Gegenstücken unterscheiden. Durch die Kombination von Einzelpartikel-Kryo-EM mit Fluoreszenz-Einzelmolekül-Analysen haben wir zwei verschiedene mitochondriale Vorinitiationsschritte charakterisiert (Khawaja et al. 2020). Unsere Ergebnisse zeigen, dass mtIF3 im Gegensatz zu allen bisher untersuchten Systemen den Aufbau des Initiationskomplexes in einer Weise aufrechterhält, die die mRNA-Bindung oder die Aufnahme der Initiator-tRNA nicht erleichtert. Darüber hinaus zeigen unsere Ergebnisse neue mitochondrienspezifische molekulare Interaktionen zwischen mitoribosomalen Proteinen und Initiationsfaktoren, die in den bisherigen Studien nicht aufgeklärt wurden, und postulieren die Mechanismen der Aktivierung der GTPase-Aktion von mtIF2. Schließlich haben wir, gestützt durch Einzelmolekülanalysen und biochemische Untersuchungen, gezeigt, dass die Translation von mitochondrienspezifischer führerloser mRNA vorzugsweise am vollen Monosom und nicht an der kleinen Untereinheit initiiert wird (Remes et al. 2022). Da unsere optimierten Protokolle, die wir für dieses Projekt entwickelt haben, eine robuste Aufreinigung von Mitoribosomen mit den dazugehörigen Faktoren ermöglichen, sind wir nun bereit, unsere Analysen zu erweitern, um andere Stadien der Translation zu beschreiben und uns dabei auf die Translationstermination und das Recycling zu konzentrieren.
2. Biogenese des Mitoribosoms
Seit Jahrzehnten wird die Ribosomenbiogenese in Bakterien und im eukaryotischen Zytosol untersucht. Dabei hat sich gezeigt, dass die Ribosomenproduktion außerordentlich komplex ist und eine große Anzahl von Hilfsfaktoren benötigt. In menschlichen Mitochondrien wurde bisher nur eine Handvoll solcher Faktoren untersucht. Unser Ziel ist es, weitere Assembly-Faktoren zu identifizieren und die Zwischenprodukte, die bei der Deletion der bereits bekannten Assembly-Faktoren entstehen, strukturell und funktionell zu charakterisieren.
Wir haben mit der Charakterisierung mehrerer GTPasen und anderer Hilfsfaktoren begonnen, die am Aufbau von Mitoribosomen beteiligt sind (z. B. GTPBP10, Busch et al. 2019; GTPBP5, Cipullo et al. 2021a). Wir ergänzen unsere biochemischen Analysen mit CryoEM, um die hochauflösenden Strukturen der Zwischenstufen des Zusammenbaus zu bestimmen (z.B. Cipullo et al.2021b; Itoh et al. 2022). Darüber hinaus interessieren wir uns für Regulationsmechanismen, die den Zusammenbau der Mitochondrien mit anderen mitochondrialen Prozessen je nach den Anforderungen der Zelle und der Umwelt koordinieren.
3. Mitochondriale RNA-bindende Proteine
In allen biologischen Systemen sind mRNAs mit RNA-bindenden Proteinen (RBPs) verbunden und bilden Komplexe, die die Genregulation von der mRNA-Synthese bis zum Zerfall steuern. Die posttranskriptionelle Regulation in Säugetiermitochondrien wird von mehreren mitochondrialen RBPs (mt-RBPs) in verschiedenen Stadien des mt-RNA-Stoffwechsels durchgeführt.
Unser Ziel ist es, neue mitochondriale Faktoren, die am RNA-Stoffwechsel und an der Translation beteiligt sind, durch biochemische, proteomische und genetische Untersuchungen zu identifizieren und anschließend die Phänotypen, die mit einer abweichenden Expression dieser Gene einhergehen, sorgfältig zu charakterisieren. In unserer Studie haben wir zum Beispiel C6orf203 als neues RNA-bindendes Protein identifiziert, das mit der großen Untereinheit des Mitoribosoms interagiert (Gopalakrishna et al. 2019). Wir ergänzen unsere biochemischen Charakterisierungen durch Strukturanalysen.
4. Mitochondriale Genexpression bei Krankheiten
Wir haben unsere mechanistischen Untersuchungen der mitochondrialen Genexpression auf Primärzellen von Patienten mit mitochondrialen Erkrankungen ausgeweitet. In den Diagnosezentren, die mit uns zusammenarbeiten, wurden kürzlich mehrere neue Kandidatengene mit pathogenen Mutationen durch Exom-Sequenzierung identifiziert. Wir führen detaillierte molekulare Analysen von Zelllinien durch, die aus solchem Patientenmaterial gewonnen wurden, um zu verstehen, wie die pathologischen Mutationen die mitochondriale Funktion beeinträchtigen und zur Krankheit führen.
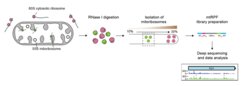
Um die Funktion der Faktoren zu untersuchen, die an mitochondrialen Krankheiten beteiligt sind, setzen wir zum Beispiel die neue Technologie des Ribosomen-Profilings ein, bei der die genauen Positionen der Ribosomen auf den Transkripten durch Nuklease-Footprinting kartiert werden. Die Tiefensequenzierung der Nuklease-geschützten Fragmente offenbart den genauen Bereich des Transkriptoms, der gerade übersetzt wird, wobei die Häufigkeit der verschiedenen Footprint-Fragmente Aufschluss über den Umfang der Übersetzung gibt. Wir haben diese Technik bereits erfolgreich angewandt, um die Wirkmechanismen verschiedener mitochondrialer Faktoren zu verstehen (Pearce et al. 2017, Gopalakrishna et al. 2019, Krüger et al. 2022) und haben die Methode für Untersuchungen an Patientenproben angepasst.